As ribozimas (palavra formada a partir de ácido ribonucleico e enzima), são RNAs dotados de atividade catalítica, principalmente de atividade RNasica, que pode ser exercida en cis, sobre eles mesmos ou en trans, sobre outras moléculas. Elas são, teoricamente, capazes de clivar um grande número de moléculas de RNA. As ribozimas representam um tipo particular e natural de moléculas antissenso com todas os atributos dos RNAs antissenso mais a vantagem da função catalítica. Estes RNAs são produzidos a partir do filamento complementar presente no lado oposto da fita codificadora que produz o RNA senso (figura abaixo). As ribozimas já foram utilizadas como RNase de restrição in vitro e como agente de controle específico da expressão genética in vivo e podem ser consideradas como ribonucleases de restrição programáveis.

Foi a partir do estudo de splicing de um RNA que teve início, por volta de 1980, a história das ribozimas. Naquela época, na Universidade do Colorado, Thomas Cech e colaboradores estavam estudando os genes do RNA ribossomal (RNAr) da Tetrahymena thermophila, um protozoário ciliado. A fim de compreender como se exprimiam esses genes, era preciso estudar o splicing e outras etapas que ocorrem durante a maturação que leva à produção do RNAr final. Esse protozoário, como outros eucariotos, possui quatro RNAr diferentes, três dos quais transcritos, a partir do DNA, em uma única molécula de RNA. Esse transcrito primário é em seguida processado e modificado, para produzir os RNAs “maduros”. Uma das primeiras etapas da maturação se efetua nos primeiros segundos que se seguem à transcrição e se trata da excisão de um íntron de aproximadamente 400 nucleotídeos, a partir de um transcrito primário de 6400 nucleotídeos. A excisão do íntron é concomitantemente com a ligação dos exons adjacentes. Thomas Cech e colaboradores constataram, para grande surpresa, que os íntrons desse RNA podiam ser excisados eficazmente in vitro, em total ausência de proteínas. Eles começaram tentando estabelecer as condições mínimas necessárias capazes de efetuar o splicing in vitro, incubando o pré-RNA ribossômico (não processado) com extratos provenientes do núcleo da Tetrahymena, que eles supunham conter as enzimas necessárias a esse processo. Thomas Cech e colaboradores perceberam que o splicing ocorria em quase todas as frações testadas, inclusive na fração controle, na qual não fora utilizada o extrato nuclear, logo em ausência de proteínas. Era preciso, entretanto, eliminar a possibilidade da existência de uma proteína contaminante ou fortemente ligada ao RNA. A confirmação de que o splicing dos pré-RNA ribossômicos da Tetrahymena é uma atividade intrínseca da estrutura do RNA e que as proteínas são “inúteis” para esse mecanismo veio da observação do splicing “espontâneo” de um RNAr sintético, produzido por transcrição in vitro, do gene correspondente clonado. O RNA produzido jamais tivera tido contato com célula, logo não podia ter sido contaminado com enzimas proteicas capazes de excisar íntrons e ligar exons. Apesar disso, ele se mostrou capaz de excisar e de processar seu próprio íntron nos mesmos locais que in vivo, em ausência total de proteína. Só a presença de magnésio e de um nucleotídeo que entra na composição do RNA, a guanosina, eram necessários. Constatou-se que a guanosina não era utilizada como fonte de energia, mas sim como um monômero a ser anexado ao íntron excisado (figura a seguir). Uma reação bioquímica, o corte da cadeia, seguido pela ligação, ocorria, portanto, sem a presença de enzima proteica. Com os íntrons do grupo I foram descobertos os primeiros RNAs catalíticos. Pela primeira vez era observado que uma macromolécula biológica, não proteica, apresentava uma atividade catalítica.

Foi constatado rapidamente que o autoexcisão se assemelhava, realmente, a uma catálise enzimática. Dentre outras propriedades, as reações apresentavam uma grande especificidade e quando o RNA era colocado em uma solução que o impedia de se dobrar, ele perdia sua capacidade de se excisar. Esta última observação é uma indicação que a estrutura tridimensional do RNA exerce um papel essencial na excisão, do mesmo modo que a estrutura terciária das cadeias dos aminoácidos intervém, de forma fundamental, na atividade enzimática das proteínas. Após os trabalhos pioneiros do grupo de Cech, uma série de outros íntrons, capazes de autoprocessamento, foram identificados em diferentes organismos. Eles têm sido descritos em outros protozoários, bactérias, fungos e organismos superiores como o milho e o feijão. Essa atividade tem sido descrita, também, em outros organoides celulares, tais como mitocôndrias e cloroplastos, não sendo, portanto, restrita aos ribossomos.
Os íntrons são classificados em dois grupos (grupo I e grupo II), com base nas suas semelhanças estruturais e no seu mecanismo de splicing. O íntron da Tetrahymena, o mais estudado e o melhor caracterizado, foi transformado em protótipo dos íntrons do grupo I (figura anterior). Os íntrons do grupo II, encontrados, geralmente, em cloroplastos de plantas e Euglena e em mitocôndrias de plantas e fungos, parecem ser menos numerosos e são menos conhecidos que os do grupo I. Ao contrário destes, a autoexcisão dos íntrons do grupo II (figura abaixo) ocorre sem guanosina e durante o processo se forma uma estrutura intermediária em forma de laço, que não é observada no caso dos íntrons da Tetrahymena ou de outros homólogos do grupo I.

Uma molécula de RNA é um longo polímero constituído de quatro nucleotídeos que são: citidina (C), uridina (U), adenosina (A) e guanosina (G). Esses quatro nucleotídeos se sucedem, segundo arranjos variados, ao longo da molécula, gerando uma sequência característica. Eles têm, dentre outras, a propriedade de se associar dois a dois por intermédio de ligações energéticas fracas do tipo pontes de hidrogênio. Esses pareamentos ocorrem entre A e U e C e G. Dessa forma, uma molécula de RNA pode se ligar a um outro RNA ou se dobrar sobre ela mesma se, neste caso, existirem sequências complementares de um lado e do outro, de modo a que os citados pareamentos sejam possíveis. Com o objetivo de conhecer precisamente as porções da molécula associadas à catálise, as sequências de nucleotídeos de diferentes íntrons do grupo I foram comparadas. Utilizando essa metodologia, F. Michel e colaboradores, do Centro de Genética Molecular de Gif-sur-Yvette na França mostraram, em 1982, que todos os íntrons do grupo I têm uma estrutura comum. Com base nesses dados foi possível determinar a posição do centro catalítico, que foi posteriormente confirmado utilizando mutações dirigidas, que consistem em modificar certos nucleotídeos dentro da cadeia. Uma outra sequência do íntron permite o reconhecimento preciso do sítio de corte ao nível da junção íntron-exon. A exemplo das reações enzimáticas clássicas, a reação de splicing é reversível, podendo os íntrons excisados reintegrar RNAs em sítios específicos. Essa reversibilidade é responsável, certamente, pela difusão dos íntrons entre os genes. É preciso mencionar que nem todos os íntrons do grupo I são capazes de autoprocessamento. Em relação a alguns, a reação é necessariamente catalisada por proteínas. Mesmo no caso de auto-catalise, sabe-se que as proteínas, de certa forma, facilitam realmente a reação, pois essa é cerca de 50 vezes mais rápida in vivo que in vitro.
Os íntrons, como descritos aqui, não se constituem verdadeiras enzimas, visto que a reação de splicing é um rearranjo intramolecular, quer dizer ela envolve um único RNA, que age sobre ele mesmo, ao contrário das enzimas clássicas que catalisam a reação de reagentes estranhos. Há ainda a considerar que nas condições mencionadas, eles não podem agir sucessivamente sobre vários RNAs. Na época da descoberta dos RNAs catalíticos, surgiu a transcrição in vitro, uma metodologia que permite a síntese de novas moléculas de RNA. Essa técnica permitiu dissecar e testar a atividade de RNAs catalíticos modificados. O resultado mais importante nesse domínio foi a obtenção de íntrons que se comportavam como verdadeiras enzimas, capazes de agir sobre outras moléculas de RNA sem ser eles mesmos modificados e serem recuperados após o processo. Isso fez nascer realmente o conceito de ribozima. Alguns anos mais tarde, Jennifer Doudna e colaboradores, do hospital geral de Massachusetts, mostraram que um íntron da Tetrahymena modificado pode se comportar como uma RNA polimerase, uma enzima proteica capaz de alongar uma molécula de RNA.
Cabe neste momento uma questão: verdadeiras enzimas RNA encontram-se nas células ou elas são apenas frutos de manipulações genéticas no laboratório? A resposta a essa questão surgiu a partir de estudos de uma enzima, a ribonuclease P (RNase P), que se encontra nas células de procariotos e de eucariotos e é responsável pela maturação do RNA transportador (RNAt). Sabe-se que os precursores do RNAt (pré RNAt) apresentam uma sequência variável na sua extremidade 5′, que deve ser cortada para dar lugar a uma molécula funcional, capaz de assumir seu papel na síntese proteica. A função da RNase P, nesse processo de maturação, é catalisar a reação de corte dessa sequência variável. Essa enzima é atípica pelo fato de ser constituída de duas subunidades, sendo uma de natureza proteica (pequena proteína de massa molecular de 20.000 daltons) e a outra é uma molécula de RNA, de 377 nucleotídeos. Admitia-se que a atividade catalítica da RNase P era dividida entre a subunidade nucleica e a subunidade proteica, esta assumindo uma atividade enzimática e o RNA um papel estrutural. Em 1983, o grupo de Sydney Altman da Universidade de Yale, em Connecticut, em colaboração com a equipe de Norman Pace, em Denver, observaram que em certas condições in vitro, o RNA dessas enzimas era capaz de efetuar a reação em questão, cortando o precursor do RNAt no lugar correto em ausência de proteínas. Estas, por outro lado, não têm ação sem o RNA. É provável, entretanto, que a proteína, desprovida, ela mesma, de atividade catalítica, ajude o RNA a funcionar nas condições naturais da célula. No ano seguinte, graças a trabalhos, ainda, realizados pelo grupo de Altman, essa hipótese de que a atividade catalítica da RNase P reside na fração nucleica foi confirmada. Dessa feita, foi efetuada a transcrição do RNA da ribonuclease P a partir de um DNA recombinante, tendo sido demonstrado que esse RNA artificial catalisava a maturação normal dos precursores do RNAt. Eliminando-se, assim, a eventualidade de uma catálise por uma proteína contaminante, essa experiência demonstrou que, pelo menos in vitro, a parte catalítica da RNase P é representada pelo RNA. In vivo, entretanto, não existe nenhuma prova que a subunidade nucleica sozinha seja capaz de catalisar a maturação dos precursores do RNAt. A subunidade proteica é certamente necessária para o funcionamento in vivo da RNase P. Ela atua aumentando a velocidade da reação, estabilizando a estrutura do RNA e modificando a especificidade da enzima. Acessoriamente, ela atua, ainda, protegendo a subunidade nucleica contra a degradação pela RNase T1, principalmente dos nucleotídeos entre as posições 170 e 270. Pela descoberta dos RNAs catalíticos Altman recebeu, juntamente com Cech, o prêmio Nobel de química 1989.
Os íntrons autocatalíticos e a RNase P não são os únicos sistemas RNA dotados de atividade catalítica. Em 1986 uma terceira categoria foi descoberta em RNAs associados a vírus de plantas, os chamados RNA satélites. Alguns vírus vegetais apresentam, além do seu próprio RNA genômico, moléculas de RNA suplementares que utilizam os mecanismos de disseminação e de multiplicação do vírus, em seu próprio benefício. Esses RNAs satélites atingem concentrações elevadas nas células infectadas e se comportam, em alguns casos, como moléculas parasitas dos vírus, atenuando a severidade dos sintomas induzidos por esses agentes infecciosos. Durante sua multiplicação, alguns desses RNA satélites se encontram sob as formas circular e linear. As cadeias lineares podem se apresentar com uma única sequência ou conter um grande número de unidades idênticas justapostas. George Bruening e colaboradores da Universidade da Califórnia, em Davis, estudaram as formas diméricas (constituídas por dois monômeros) do RNA satélite de um vírus parasita do tabaco. Eles mostraram, em 1986, que essas moléculas diméricas podiam catalisar sua própria clivagem, liberando os dois RNAs satélites monoméricos.
By:
Blog do Prof. Djalma Santos
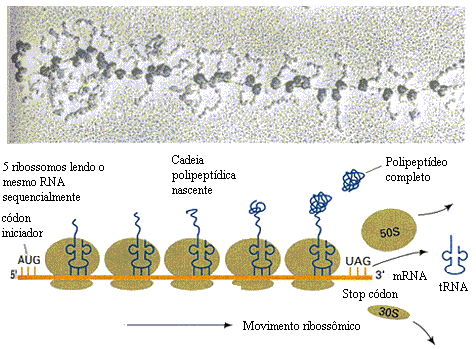

 A toxicidade da planta é conhecida desde tempos remotos. Segundo Lord et al.(1994), há mais de um século atrás foi isolada das sementes da mamona uma proteína denominada ricina. Nesta época, acreditava-se que a toxicidade desta proteína resultava de sua habilidade de aglutinar, in vitro, células vermelhas do sangue. Estudos mais recentes mostraram que as preparações de ricina daquela época eram, na verdade, uma mistura de uma potente citotoxina, a ricina, e uma hematoaglutinina, a Ricinus communis aglutinina (RCA). Porém, sabe-se que esta hematoaglutinina é oralmente inativa, e só apresenta ação aglutinante de hemácias in vitro, ou quando administrada intravenosamente (Lampe, 1991). Assim, descarta-se a hipótese da intoxicação ser causada por aglutinação de eritrócitos.Vários trabalhos tentaram elucidar a ação da ricina em células animais. Foi assim que em 1988, Endo & Tsurugi divulgaram um trabalho decisivo, no qual descreveram o mecanismo da ação catalítica da ricina na unidade 60S dos ribossomos das células eucarióticas. Após a divulgação deste trabalho, várias proteínas estrutural e funcionalmente relacionadas à ricina foram descritas para uma grande variedade de plantas superiores. Estas proteínas, juntamente com a ricina, formam um grupo e são coletivamente conhecidas como “proteínas inativadoras de ribossomos” (RIPs). Estas enzimas inativam especificamente e irreversivelmente ribossomos eucarióticos, impedindo a síntese protéica. Elas podem ocorrer como monômeros de aproximadamente 30kDa (chamadas RIPS tipo I), ou, em certos tecidos vegetais, como um heterodímero, no qual uma RIP tipo I está covalentemente unida através de uma ponte dissulfeto a um segundo polipetídeo, cuja massa também está em torno de 30 kDa. Este segundo polipetídeo é descrito como uma lectina ligadora de galactose, e o heterodímero formado é chamado de RIP tipo II (Lord et al., 1994).
A toxicidade da planta é conhecida desde tempos remotos. Segundo Lord et al.(1994), há mais de um século atrás foi isolada das sementes da mamona uma proteína denominada ricina. Nesta época, acreditava-se que a toxicidade desta proteína resultava de sua habilidade de aglutinar, in vitro, células vermelhas do sangue. Estudos mais recentes mostraram que as preparações de ricina daquela época eram, na verdade, uma mistura de uma potente citotoxina, a ricina, e uma hematoaglutinina, a Ricinus communis aglutinina (RCA). Porém, sabe-se que esta hematoaglutinina é oralmente inativa, e só apresenta ação aglutinante de hemácias in vitro, ou quando administrada intravenosamente (Lampe, 1991). Assim, descarta-se a hipótese da intoxicação ser causada por aglutinação de eritrócitos.Vários trabalhos tentaram elucidar a ação da ricina em células animais. Foi assim que em 1988, Endo & Tsurugi divulgaram um trabalho decisivo, no qual descreveram o mecanismo da ação catalítica da ricina na unidade 60S dos ribossomos das células eucarióticas. Após a divulgação deste trabalho, várias proteínas estrutural e funcionalmente relacionadas à ricina foram descritas para uma grande variedade de plantas superiores. Estas proteínas, juntamente com a ricina, formam um grupo e são coletivamente conhecidas como “proteínas inativadoras de ribossomos” (RIPs). Estas enzimas inativam especificamente e irreversivelmente ribossomos eucarióticos, impedindo a síntese protéica. Elas podem ocorrer como monômeros de aproximadamente 30kDa (chamadas RIPS tipo I), ou, em certos tecidos vegetais, como um heterodímero, no qual uma RIP tipo I está covalentemente unida através de uma ponte dissulfeto a um segundo polipetídeo, cuja massa também está em torno de 30 kDa. Este segundo polipetídeo é descrito como uma lectina ligadora de galactose, e o heterodímero formado é chamado de RIP tipo II (Lord et al., 1994). Como monômeros, as RIPs não são citotóxicas, pois não atravessam a membrana celular eucariótica. Na verdade, certos tecidos vegetais ricos em RIPs tipo I, como o germe de trigo e o grão de cevada, são largamente consumidos por seres humanos e animais sem nenhum dano celular. Porém, RIPs tipo II ligam-se às células eucarióticas através de interações com galactosídeos da superfície celular e, após subseqüente entrada no citosol, promovem a morte celular por inibição da síntese de proteínas (Lord et al., 1994).
Como monômeros, as RIPs não são citotóxicas, pois não atravessam a membrana celular eucariótica. Na verdade, certos tecidos vegetais ricos em RIPs tipo I, como o germe de trigo e o grão de cevada, são largamente consumidos por seres humanos e animais sem nenhum dano celular. Porém, RIPs tipo II ligam-se às células eucarióticas através de interações com galactosídeos da superfície celular e, após subseqüente entrada no citosol, promovem a morte celular por inibição da síntese de proteínas (Lord et al., 1994).



